
-

生物通官微
陪你抓住生命科技
跳动的脉搏
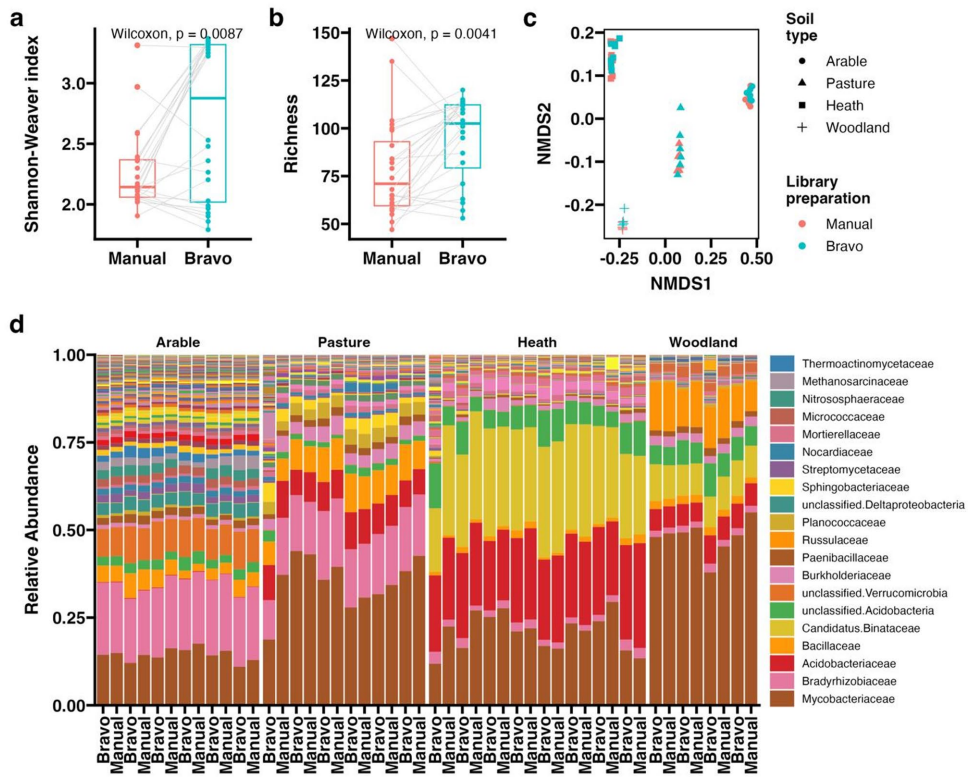
牛津纳米孔长读长测序在环境宏基因组学中的自动化应用:提升效率与重现性的关键研究
【字体: 大 中 小 】 时间:2025年09月27日 来源:BMC Genomics 3.7
编辑推荐:
本研究针对环境宏基因组学中长读长测序(ONT)自动化建库流程的验证需求,通过对比手动与自动化(Bravo平台)方法,证实自动化建库在保持微生物群落结构一致性的同时,提升了稀有物种检出率和分类效率,为高通量、高重现性宏基因组研究提供了关键技术支持。

 生物通微信公众号
生物通微信公众号
知名企业招聘